16.2: Аналіз послідовності
- Page ID
- 6530
\( \newcommand{\vecs}[1]{\overset { \scriptstyle \rightharpoonup} {\mathbf{#1}} } \) \( \newcommand{\vecd}[1]{\overset{-\!-\!\rightharpoonup}{\vphantom{a}\smash {#1}}} \)\(\newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\) \( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\) \( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\) \( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\) \( \newcommand{\Span}{\mathrm{span}}\) \(\newcommand{\id}{\mathrm{id}}\) \( \newcommand{\Span}{\mathrm{span}}\) \( \newcommand{\kernel}{\mathrm{null}\,}\) \( \newcommand{\range}{\mathrm{range}\,}\) \( \newcommand{\RealPart}{\mathrm{Re}}\) \( \newcommand{\ImaginaryPart}{\mathrm{Im}}\) \( \newcommand{\Argument}{\mathrm{Arg}}\) \( \newcommand{\norm}[1]{\| #1 \|}\) \( \newcommand{\inner}[2]{\langle #1, #2 \rangle}\) \( \newcommand{\Span}{\mathrm{span}}\)
- Скачайте файл Lacz.gb і відкрийте в текстовому редакторі.
- Це файл формату Genbank, який містить послідовність, наступну за словом «ORIGIN» і закінчується на «//».
- Перед послідовністю йде партія описової інформації, включаючи ідентифікатори посилань, організмів та бази даних перехресних посилань. Хоча це не означає для вас багато, відповідну базу даних в Genbank можна запитати, щоб розкрити більше інформації про послідовність.
- Скачайте файл LACZ.fasta і відкрийте в текстовому редакторі (Блокнот).
- Зверніть увагу на просту структуру файлу fasta, що починається з «>» та опису послідовності.
- Це послідовність ДНК. Але ДНК, як правило, дволанцюгова! Можна припустити послідовність другої пасма, оскільки вона буде доповнювати цю.
- За угодою: ми знаємо, що ця послідовність становить 5 ′ → 3′.
- Цей текст містить частину генома кишкової палички, яка включає ген під назвою LaCZ.
- Цей файл не містить жодної анотації, яка вказує, де насправді починається або закінчується послідовність генів.
- Запустіть UGENE і відкрийте обидва файли. Вони з'являться в лівій частині панелі «Об'єкти».
- На дисплеї за замовчуванням автоматично відображається зворотне доповнення ланцюга ДНК та всіх 6 відкритих кадрів читання (ORF).
- Щоб спростити вигляд, натисніть на 'C', щоб видалити комплементарну пасмо (дивіться курсор на зображенні).
4. Підрахуйте ORF:
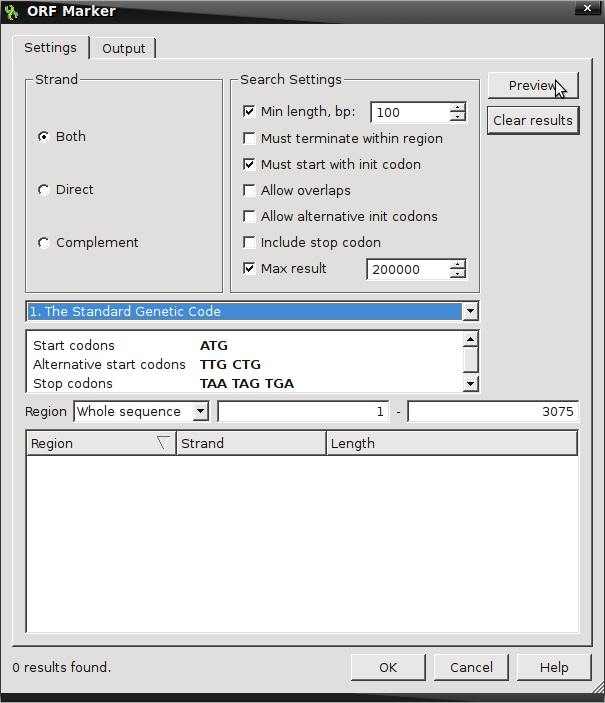
- Знайдіть ORF, клікнувши правою кнопкою миші по послідовності і виберіть «Аналіз → Знайти ORF»
- Налаштування за замовчуванням шукає ORF на обох нитках з мінімальною довжиною 100 нуклеотидів
- Відкрите читання кадру тут визначається як щось, що починається з ініціації або запуску кодонів зі Стандартного генетичного коду (ATG) і двох додаткових альтернативних початкових кодонів (TTG & CTG), що завершується будь-яким з трьох стандартних стоп-кодонів ( ТАА, ТЕГ, ТЕГ)
- Вибір пункту «Попередній перегляд» надасть кількість можливих ORF, які відповідають цим критеріям.
5. Двічі клацніть на Lacz.gb на панелі «Об'єкти», щоб активувати перегляд.
- Цей файл тепер показує ту саму послідовність з інформацією про ДНК.

- Розгорніть різні функції на панелі «Анотації» внизу, щоб вивчити функції послідовності.



