11.6: Використання R для аналізу основних компонентів
- Page ID
- 17721
Щоб проілюструвати, як ми можемо використовувати R для завершення кластерного аналізу: скористайтеся цим посиланням та збережіть файл allSpec.csv у вашій робочій директорії. Дані в цьому файлі складаються з 80 рядків і 642 стовпців. Кожен ряд є незалежним зразком, який містить один або кілька наступних катіонів перехідних металів: Cu 2 +, Co 2 +, Cr 3 + і Ni 2 +. У перших семи графах представлена інформація про зразки:
- id зразка (у вигляді custd_1 для єдиного стандарту Cu 2 + або nicu_mix1 для суміші Ni 2 + і Cu 2 +)
- перелік аналітів у зразку (у вигляді cuco для зразка, що містить Cu 2 + і Co 2 +)
- кількість аналітів у зразку (число від 1 до 4 і марковані як розміри)
- молярна концентрація Cu 2 + в зразку
- молярна концентрація Co 2 + в зразку
- молярна концентрація Cr 3 + в зразку
- молярна концентрація Ni 2 + в зразку
Решта стовпців містять значення поглинання на 635 довжині хвиль між 380,5 нм і 899,5 нм.
По-перше, нам потрібно прочитати дані в R, що ми робимо за допомогою функції read.csv ()
Спец_дані <- read.csv (» allSpec.csv «, перевірка.імена = ПОМИЛКОВО)
де параметр check.names = FALSE перевизначає типову функцію, щоб не дозволити ім'я стовпця починатися з числа. Далі ми створимо підмножину цього великого набору даних для роботи з
довжина хвилі_іди = seq (8, 642, 40)
sample_ids = c (1, 6, 11, 21:25, 38:53)
pca_data = spec_data [sample_ids, довжина хвилі_ids]
де wavelength_ids - вектор, який ідентифікує 16 однаково розташованих довжин хвиль, sample_ids - це вектор, який ідентифікує 24 зразки, які містять один або кілька катіонів Cu 2 +, Co 2 + та Cr 3 +, і cluster_data - це кадр даних, який містить значення поглинання для цих 24 зразків на цих 16 довжині хвиль.
Для завершення аналізу основних компонентів ми будемо використовувати R функцію prcomp (), яка приймає загальний вигляд
prcomp (об'єкт, центр, масштаб)
де об'єкт - це кадр даних або матриця, яка містить наші дані, а центр та масштаб - це логічні значення, які вказують, чи слід спочатку центрувати та масштабувати дані, перш ніж завершити аналіз. Коли ми центруємо та масштабуємо наші дані, кожна змінна (в даному випадку поглинання на кожній довжині хвилі) регулюється таким чином, щоб її середнє значення дорівнювало нулю, а дисперсія - одна. Це призводить до розміщення всіх змінних у загальній шкалі, що гарантує, що будь-яка різниця у відносній величині змінних не впливає на аналіз основного компонента.
pca_results = prcomp (pca_data, центр = ІСТИНА, масштаб = ІСТИНА)
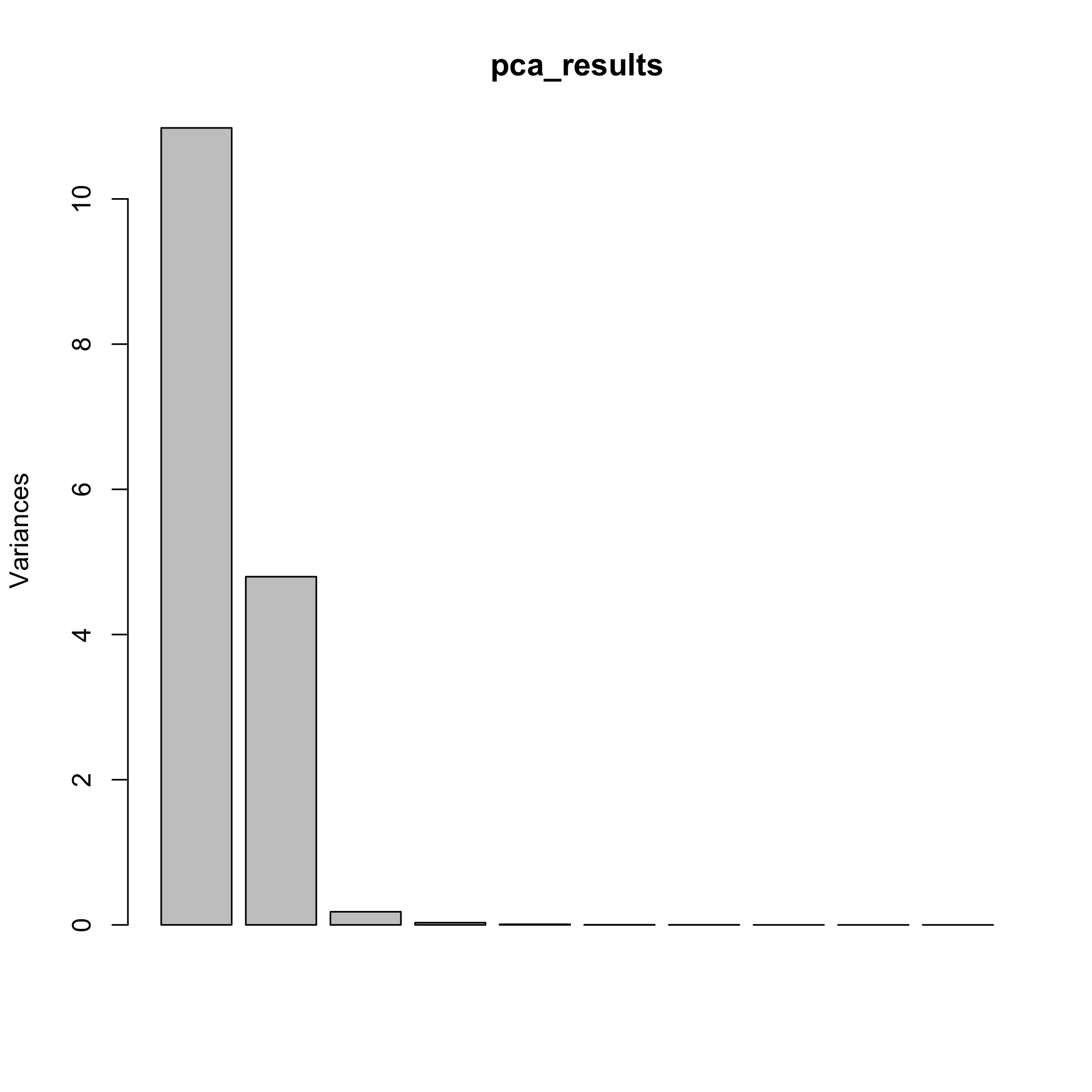
Функція prcomp () повертає різноманітну інформацію, яку ми можемо використовувати для вивчення результатів, включаючи стандартне відхилення для кожного основного компонента, sdev, матрицю з навантаженнями, обертання, матрицю з оцінками, x та значення, які використовуються для центрувати і масштабувати вихідні дані. Наприклад, функція summary () повертає стандартні відхилення для та частку загальної дисперсії, поясненої кожним основним компонентом, а також сукупну частку дисперсії, пояснену основними компонентами.
резюме (pca_результати)
Важливість компонентів:
ПК1 ШК2 ПК3 ПК4 ПК5 Ш-6 Ш/7 ПК8 Ш-9
Стандартне відхилення 3.3134 2.1901 0,42561 0,17585 0,09384 0,0407 0,04026 0,01253 0,01049
Частка дисперсії 0,6862 0,2998 0,01132 0,00193 0.00055 0.00013 0.00010 0.00001
Сукупна пропорція 0,6862 0,9859 0,99725 0,99919 0,99974 0,99987 0,99997 0,99998 0,9999
ПК/10 шт 11 шт 12 шт 13 шт 14 шт 15 шт 16
Стандартне відхилення 0,009211 0,007084 0,004478 0,00416 0,003039 0,002377 0,001504
Частка дисперсії 0,000010 0,000000 0,00000 0,00000 0,00000 0,000000
Сукупна пропорція 0.99990 1.000000 1.000000 1.000000 1.000000 1.000000 1.000000
Ми також можемо дослідити дисперсію кожної головної складової (квадрат її стандартного відхилення) у вигляді стовпчастої графіки, передаючи результати аналізу головної складової функції plot ().
сюжет (pca_результати)
Як зазначалося вище, 24 зразки включають один, два або три з катіонів Cu 2 +, Co 2 + і Cr 3 +, що відповідає нашим результатам, якщо окремі рішення зроблені шляхом об'єднання разом аліквот вихідних розчинів Cu 2 +, Co 2 +, і Cr 3 + і розведення до загального обсягу. При цьому обсяг вихідного розчину для одного катіону встановлює обмеження на обсяги інших катіонів таким чином, що трикомпонентна суміш по суті має дві незалежні змінні.
Щоб вивчити бали для аналізу основних компонентів, ми передаємо бали у функцію plot (), тут використовуючи pch = 19, щоб відобразити їх як заповнені точки.
сюжет (pca_результати $x, pch = 19)
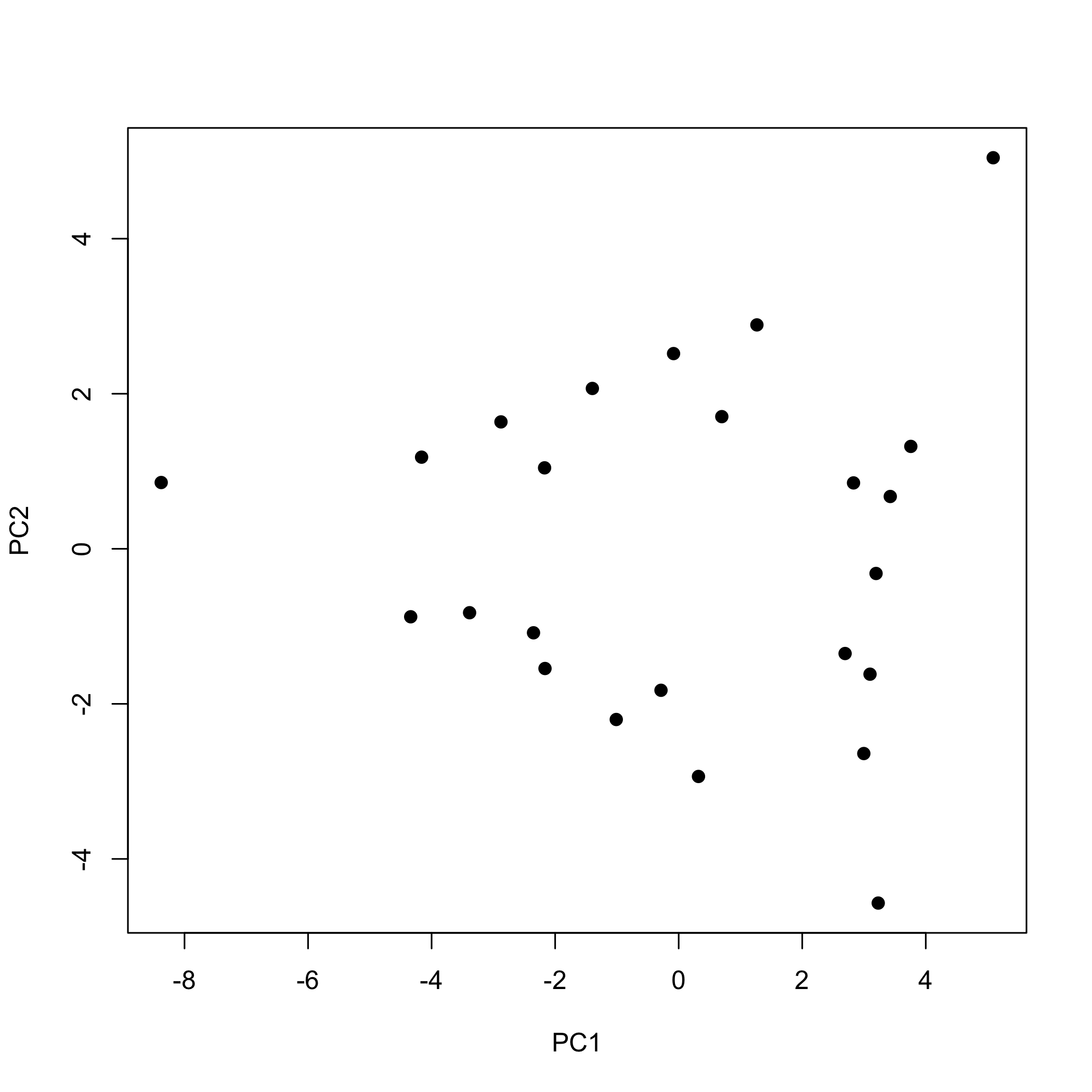
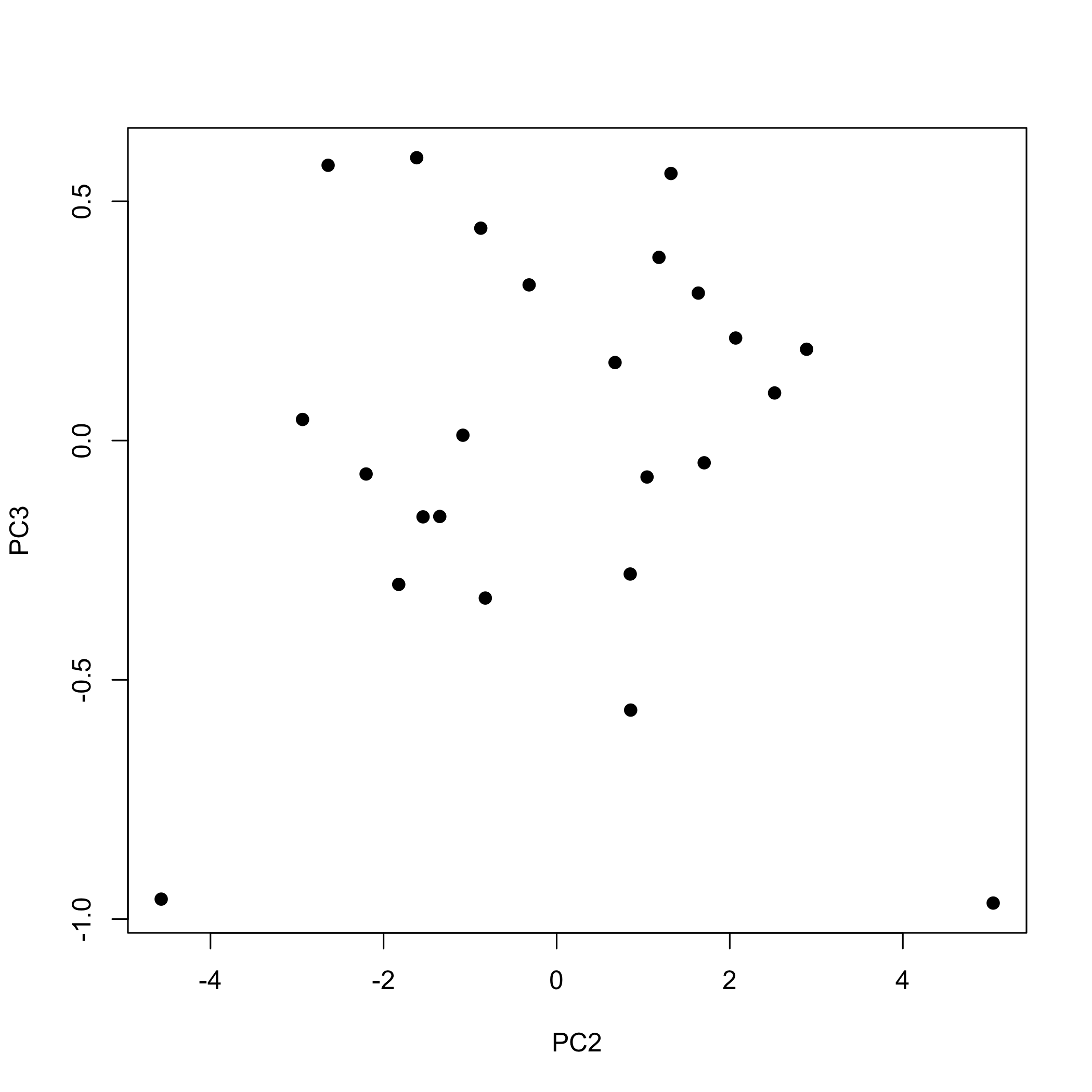
За замовчуванням функція plot () відображає значення для перших двох основних компонентів, причому перший (PC1) розміщений на осі x, а другий (PC2) розміщений на осі y. Якщо ми хочемо вивчити інші основні компоненти, то ми повинні вказати їх при виклику функції plot (); наступна команда, наприклад, використовує оцінки для другого та третього основних компонентів.
сюжет (x = pca_result$ х [2], y = pca_result$ х [3], pch = 19, xlab = «PC2", ylab = «PC3")
Якщо ми хочемо відобразити перші три основні компоненти за допомогою одного і того ж графіка, то ми можемо використовувати функцію Scatter3D () з пакету plot3D, яка приймає загальний вигляд
бібліотека (сюжет 3D)
розсіювання3D (x = pca_result$ x [,1], y = pca_results$ х [2], z = pca_results$ x [,3], pch = 19, тип = «h», тета = 25, phi = 20, тип кліща = «детальний», колвар = NULL)
де ми використовуємо функцію library () для завантаження пакунка в наш сеанс R (примітка: це припускає, що ви встановили пакет plot3D). Тип опції = «h» скидає горизонтальну лінію від кожної точки вниз до площини для PC1 і PC2, що допомагає нам зорієнтувати точки в просторі. За замовчуванням графік використовує колір, щоб показати значення кожної точки третього основного компонента (відображається на осі z); тут ми встановлюємо colvar = NULL, щоб відобразити всі точки одним кольором.
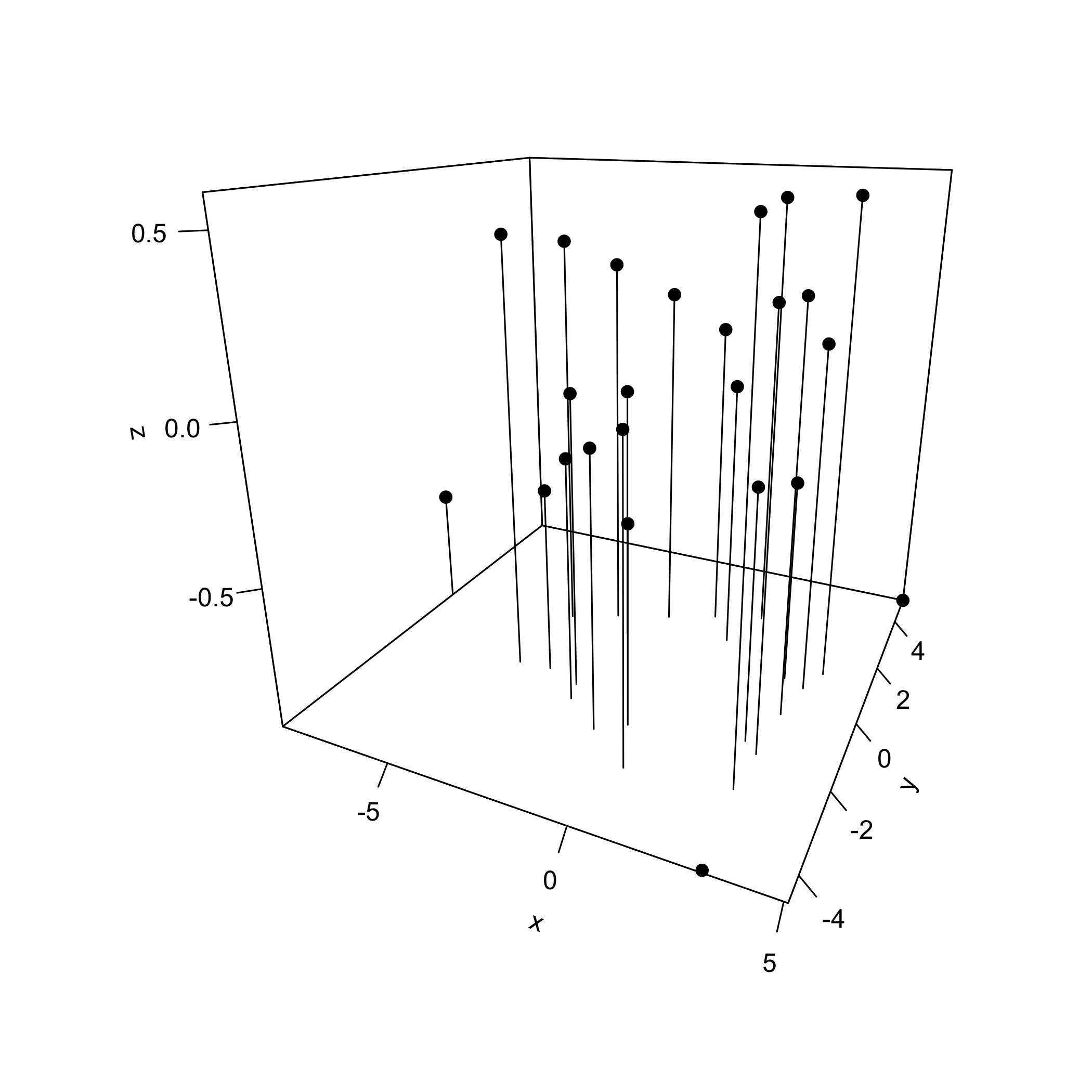
Хоча графіки тут не показані, ми можемо використовувати ті ж команди, замінивши x обертанням, для відображення навантажень.
сюжет (pca_results $ обертання, pch = 19)
сюжет (x = pca_results$ обертання [2], y = pca_result$ обертання [,3], pch = 19, xlab = «PC2", ylab = «PC3")
Розкид 3D (x = pca_results $ обертання [1], y = pca_result$ обертання [2], z = pca_results$ обертання [,3], pch = 19, тип = «h», тета = 25, phi = 20, тип кліща = «детальний», колвар = NULL)
Іншим способом перегляду результатів аналізу основних компонентів є відображення балів та навантажень на одному графіку, що ми можемо зробити за допомогою функції biplot ().
біплот (pca_результати, cex = c (2, 0.6), xlabs = rep («•», 24)
де опція xlabs = rep («•», 24) перевизначає функцію за замовчуванням для відображення балів у вигляді чисел, замінюючи їх крапками, а cex = c (2, 0.6) використовується для збільшення розміру точок і зменшення розміру міток для завантажень.
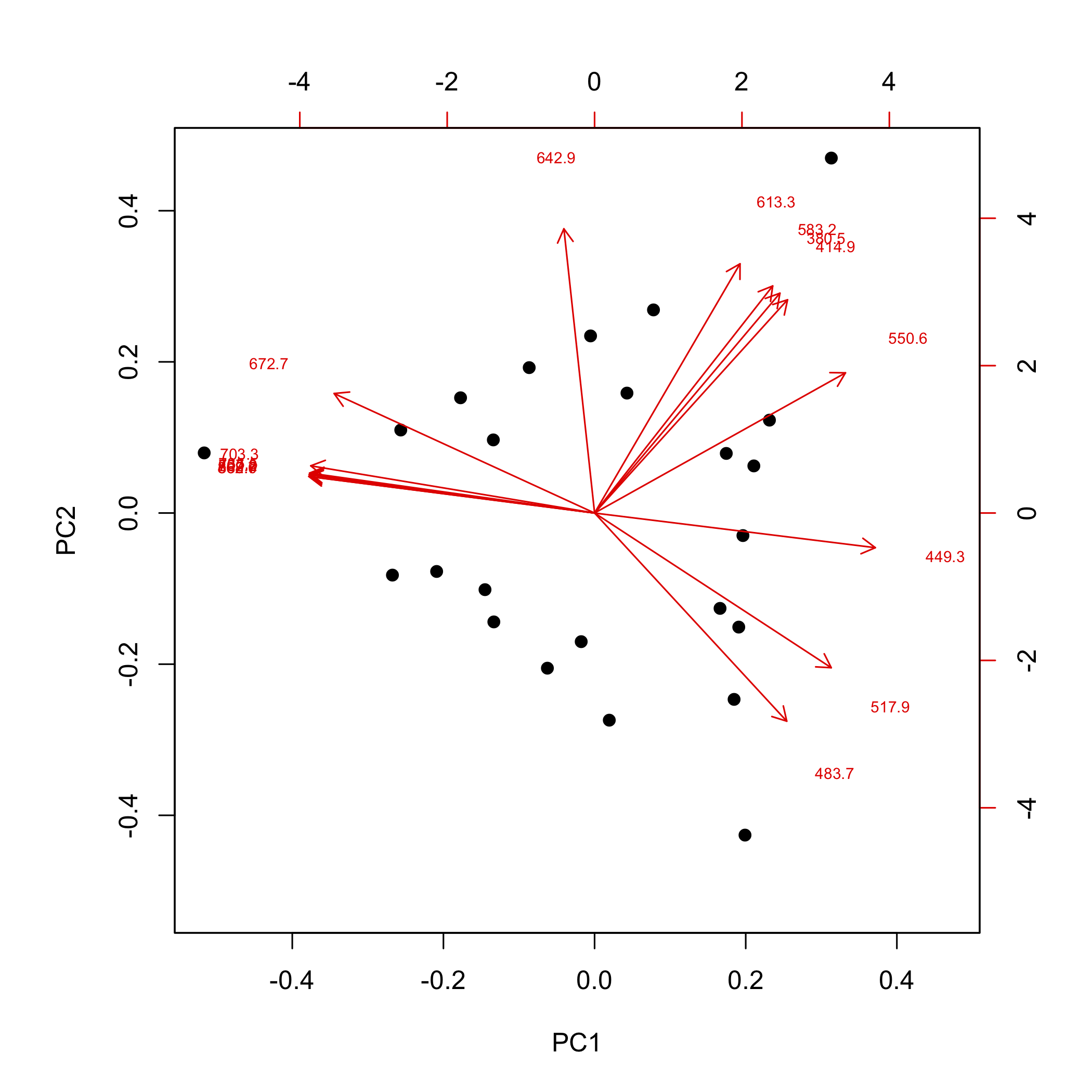
У цьому бісюжеті оцінки відображаються у вигляді точок, а навантаження відображаються у вигляді стрілок, які починаються з початку та вказують на окремі навантаження, які позначаються довжинами хвиль, пов'язаними з навантаженнями. Для цього набору даних бали та навантаження, які спільно розташовані один з одним, представляють зразки та довжини хвиль, які сильно корелюють між собою. Наприклад, зразок, бал якого знаходиться у верхньому правому куті, сильно пов'язаний з поглинанням світла з довжинами хвиль 613,3 нм, 583,2 нм, 380,5 нм і 414,9 нм.
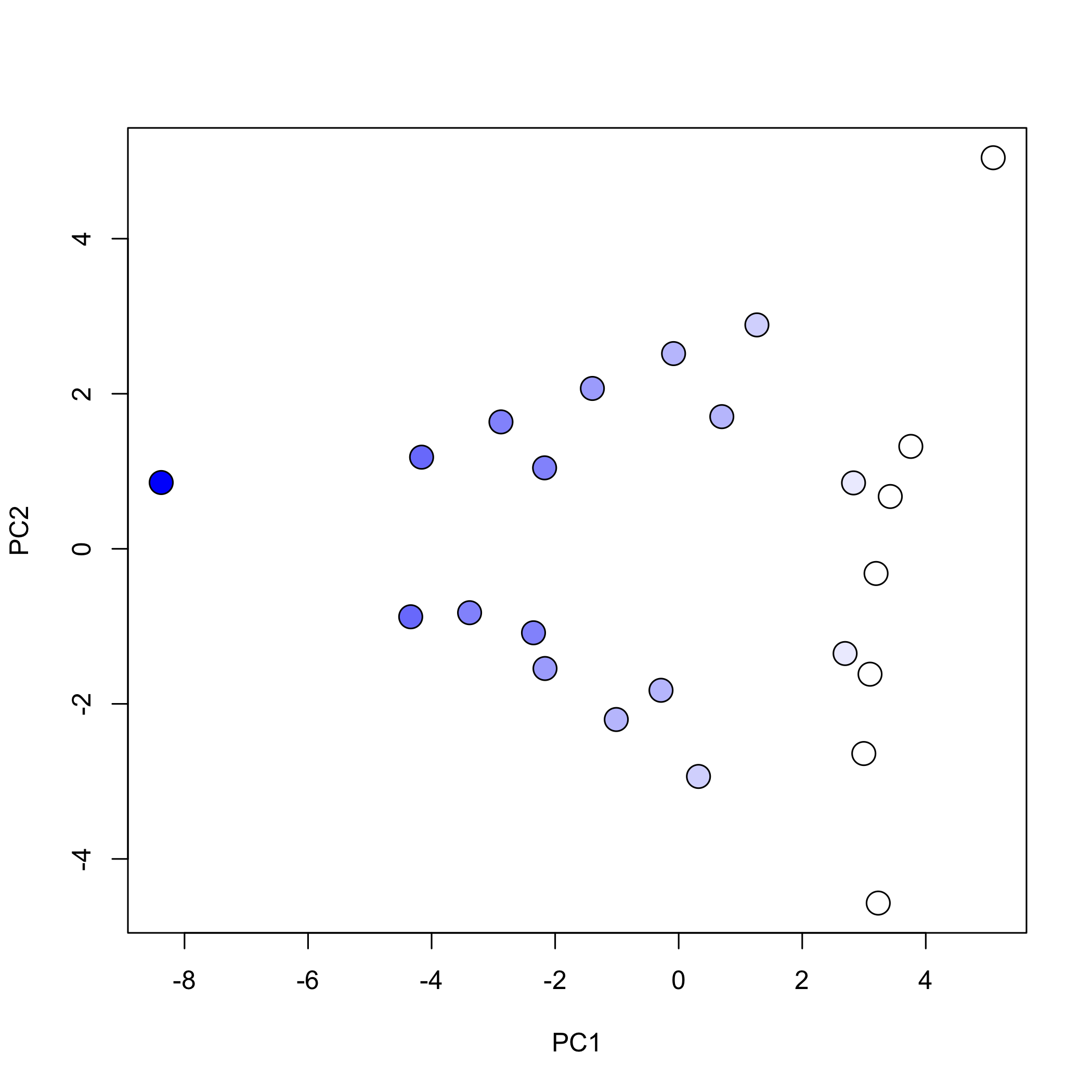
Нарешті, ми можемо використовувати колір для виділення функцій з нашого набору даних. Наприклад, наступні рядки коду створюють графік балів, який використовує палітру кольорів для позначення відносної концентрації Cu 2 + у зразку.
cu_palette = Колірна палітра (c («білий», «синій»))
cu_color = cu_pallete (50) [як числовий (вирізати (Spec_data$conccu [sample_ids], перерви = 50))]
Функція ColorRampPalette () приймає вектор кольорів - у цьому випадку білий і синій - і повертає функцію, яку ми можемо використовувати для створення палітри кольорів, яка проходить від чистого білого до чистого синього. Потім ми використовуємо цю функцію для створення 50 відтінків білого і синього.
палітра (50)
[1] "#FFFFFF" "#F9F9FF" #F4F4FF "#EFEFFF" "#EAEAFF" "#E4E4FF" "#DFDFFF" "#DADAFF"
[9] "#D5D5FF" "#D0D0FF" #CACAFF "#C5C5FF" "#C0C0FF" "#BBBBFF" "#B6B6FF" "#B0B0FF"
[17] "#ABABFF" "#A6A6FF" #A1A1FF "#9C9CFF" "#9696FF" "#9191FF" "#8C8CFF" "#8787FF"
[25] "#8282FF" "#7C7CFF" #7777FF "#7272FF" "#6D6DFF" "#6868FF" "#6262FF" "#5D5DFF"
[33] "#5858FF" "#5353FF" #4E4EFF "#4848FF" "#4343FF" "#3E3EFF" "#3939FF" "#3434FF"
[41] "#2E2EFF" "#2929FF" #2424FF "#1F1FFF" "#1A1AFF" "#1414FF" "#0F0FFF" "#0A0AFF"
[49] "#0505FF" #0000FF "
де #FFFFFF - шістнадцятковий код для чистого білого кольору, а #0000FF - шістнадцятковий код для чистого синього кольору. Остання частина цього рядка коду
cu_color = cu_pallete (50) [як.числовий (вирізати (Spec_data$Conccu [вибірки_ідентифікатори], перерви = 50))]
отримує концентрації міді в кожному з наших 24 зразків і призначає шістнадцятковий код для відтінку синього, який вказує на відносну концентрацію міді в зразку. Тут ми бачимо, що перший зразок має шістнадцятковий код #0000FF для чистого синього кольору, що означає, що цей зразок має найбільшу концентрацію міді, а зразки 2-8 мають гексадемічні коди #FFFFFF для чистого білого кольору, що означає, що ці зразки не містять жодної міді.
cu_color
[1] "#0000FF" "#FFFFFF" #FFFFFF "#FFFFFF" "#FFFFFF" "#FFFFFF" "#FFFFFF" "#FFFFFF"
[9] "#D0D0FF" "#B6B6FF" #9C9CFF "#8282FF" "#6868FF" "#D0D0FF" "#B6B6FF" "#9C9CFF"
[17] "#8282FF" "#6868FF" #EAEAFF "#EAEAFF" "#B6B6FF" "#B6B6FF" "#8282FF" "#8282FF"
Нарешті, ми створюємо графік балів, використовуючи pch = 21 для відкритого кола, колір фону якого ми позначаємо за допомогою bg = cu_color і де ми використовуємо cex = 2 для збільшення розміру точок.
сюжет (pca_result$ x, pch = 21, bg = cu_color, секс = 2)