11.5: Використання R для кластерного аналізу
- Page ID
- 17759
Щоб проілюструвати, як ми можемо використовувати R для завершення кластерного аналізу: скористайтеся цим посиланням та збережіть файл allSpec.csv у вашій робочій директорії. Дані в цьому файлі складаються з 80 рядків і 642 стовпців. Кожен ряд є незалежним зразком, який містить один або кілька наступних катіонів перехідних металів: Cu 2 +, Co 2 +, Cr 3 + і Ni 2 +. У перших семи графах представлена інформація про зразки:
- id зразка (у вигляді custd_1 для єдиного стандарту Cu 2 + або nicu_mix1 для суміші Ni 2 + і Cu 2 +)
- перелік аналітів у зразку (у вигляді cuco для зразка, що містить Cu 2 + і Co 2 +)
- кількість аналітів у зразку (число від 1 до 4 і марковане як розміри)
- молярна концентрація Cu 2 + в зразку
- молярна концентрація Co 2 + в зразку
- молярна концентрація Cr 3 + в зразку
- молярна концентрація Ni 2 + в зразку
Решта стовпців містять значення поглинання на 635 довжині хвиль між 380,5 нм і 899,5 нм.
По-перше, нам потрібно прочитати дані в R, що ми робимо за допомогою функції read.csv ()
Спец_дані <- read.csv (» allSpec.csv «, перевірка.імена = ПОМИЛКОВО)
де параметр check.names = FALSE перевизначає типове значення функції, щоб не дозволити ім'я стовпця починатися з числа. Далі ми створимо підмножину цього великого набору даних для роботи
довжина хвилі_іди = seq (8, 642, 40)
sample_ids = c (1, 6, 11, 21:25, 38:53)
кластер_дані = спец_дані [sample_ids, довжина хвилі_ids]
де wavelength_ids - вектор, який ідентифікує 16 однаково розташованих довжин хвиль, sample_ids - це вектор, який ідентифікує 24 зразки, які містять один або кілька катіонів Cu 2 +, Co 2 + та Cr 3 +, і cluster_data - це кадр даних, який містить значення поглинання для цих 24 зразків на цих 16 довжині хвиль.
Перш ніж ми зможемо завершити кластерний аналіз, ми спочатку повинні обчислити відстань між\(24 \times 16 = 384\) точками, що складають наші дані. Для цього використовуємо функцію dist (), яка приймає загальний вигляд
dist (об'єкт, метод)
де об'єкт - це кадр даних або матриця з нашими даними. Існує ряд варіантів методу, але ми будемо використовувати за замовчуванням, який є евклідовим.
cluster_dist = дист (кластер_дані, метод = «евклідовий»)
кластер_dist
1 6 11 21 22 23 24 25
6 1,533 28104
1 1,73128979 0.96493008
21 1,48359716 0,249 7370 0.77766228
2 1,49208058 0.32863786 0,6885 2029 0.09664215
23 1,49457333 0,42903074 0.57499 0.21089686 0.11755129
24 1.51211374 0.52218072 0,47 457024 0.31016429 0.21830998 0.10205547
25 1.55862311 0.61154277 0.39798649 0.39406580 0.30194838 0.191251 0,097 1283
38 1.17069314 0.38098750 0.96982420 0.34254297 0.38830178 0.45418483 0.53114050 0.61729900
Тут показано лише невелику частину значень у cluster_dist; кожен запис показує відстань між двома із 24 зразків.
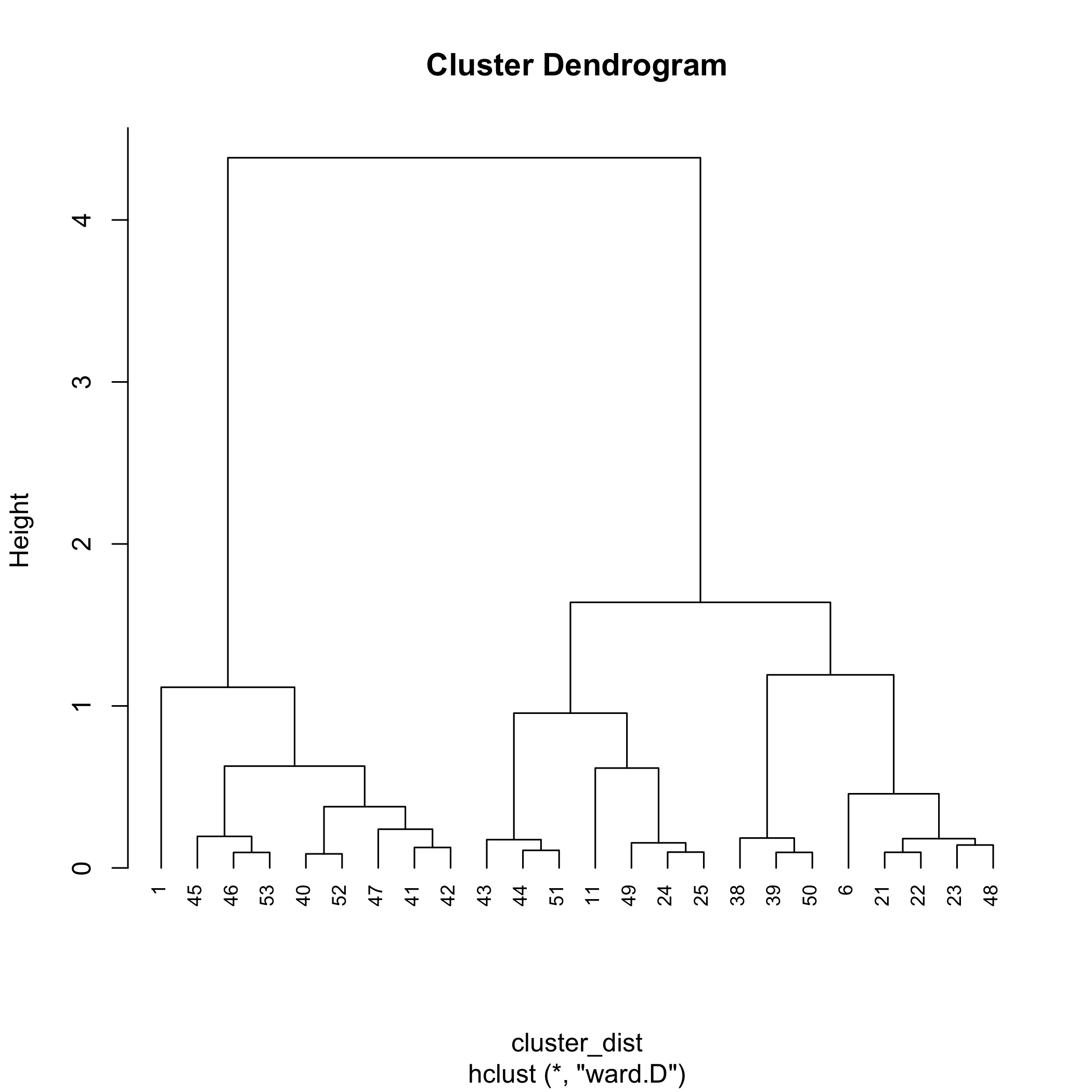
При обчисленні відстаней ми можемо використовувати функцію hclust () R для завершення кластерного аналізу. Загальна форма функції
hclust (об'єкт, метод)
де object — результат, створений за допомогою dist (), який містить відстані між точками. Існує ряд варіантів для методу - тут ми використовуємо метод Ward.d - збереження вихідних даних до об'єкту cluster_results, щоб ми мали доступ до результатів.
cluster_results = скупчення (кластер_дист, метод = «Уорд.»)
Для перегляду кластерної діаграми передаємо об'єкт cluster_results у функцію plot (), де hang = -1 розширює кожну вертикальну лінію на висоту нуля. За замовчуванням мітки внизу дендрограми є ідентифікаторами зразків; cex коригує розмір цих міток.
сюжет (кластер_результати, зависання = -1, cex = 0,75)
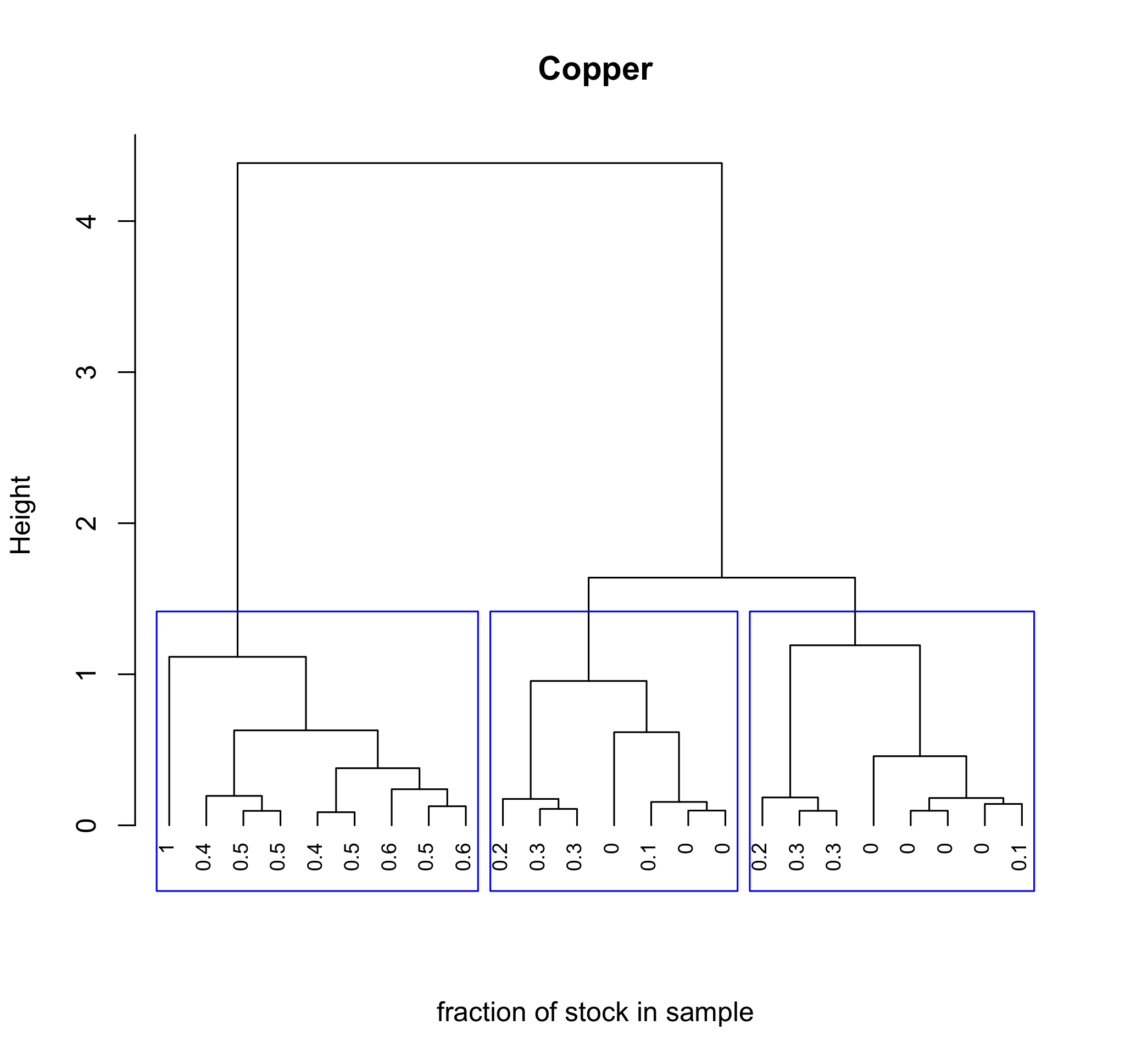
За допомогою декількох рядків коду ми можемо додати корисні деталі до нашого сюжету. Тут, наприклад, ми визначаємо частку вихідного розчину Cu 2 + у кожному зразку і використовуємо ці значення як мітки, а 24 вибірки розділимо на три великі кластери за допомогою функції rect.clust (), де k - кількість кластерів для виділення та який вказує, який з цих кластерів відображати за допомогою прямокутної коробки.
cluster_copper = spec_data$Conccu/spec_data$Conccu [1]
сюжет (cluster_results, зависання = -1, мітки = cluster_copper [sample_ids], основний = «Мідь», xlab = «частка запасу в зразку», sub = «», cex = 0.75)
прямотий.hclust (cluster_results, k = 3, який = c (1) 2,3), межа = «синій»)
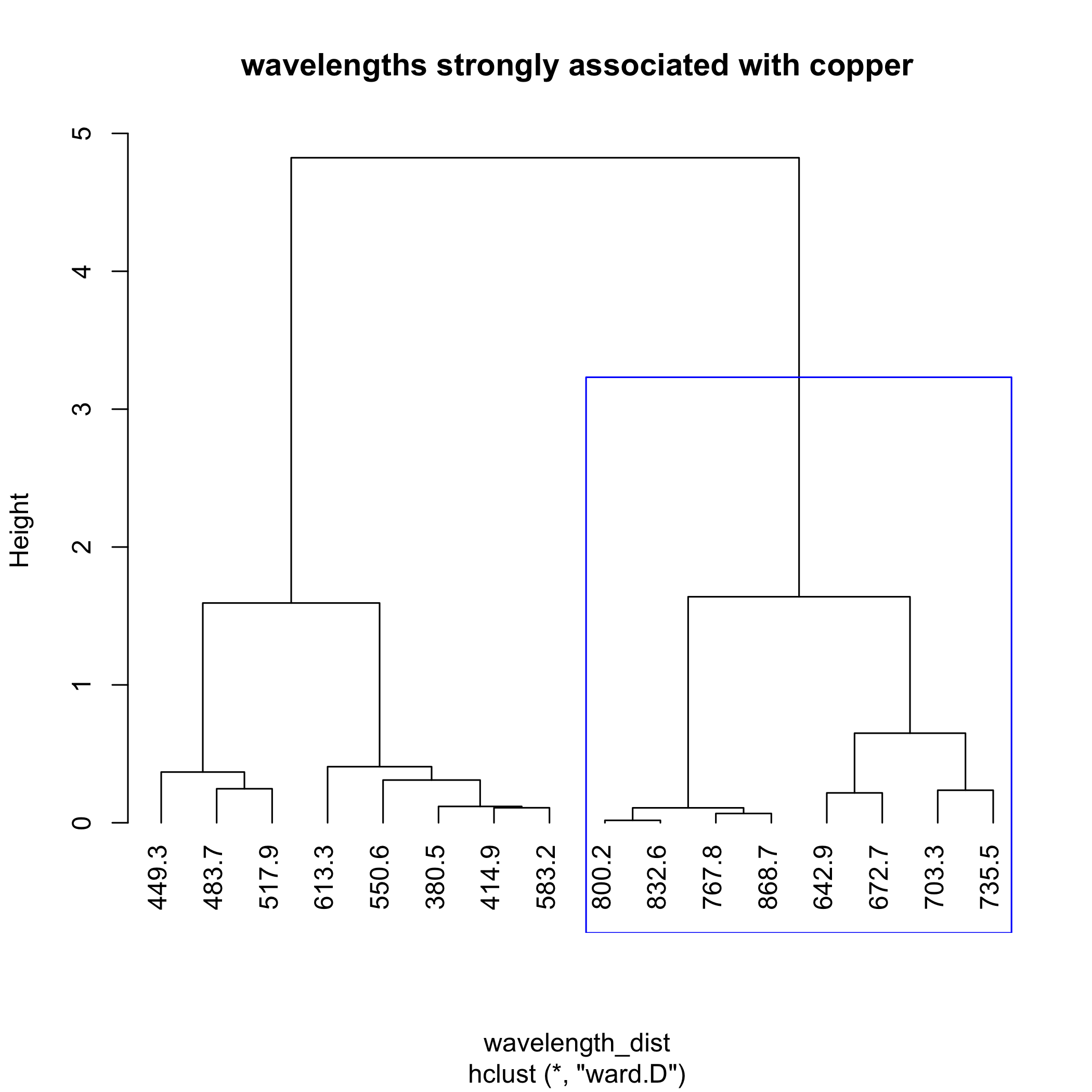
Наступний код показує, як ми можемо використовувати той самий набір даних з 24 зразків і 16 довжини хвилі для завершення кластерної діаграми для довжин хвиль. Використання функції t () у функції dist () приймає транспонування наших даних так, що рядки мають довжину хвиль 16, а стовпці - зразки 24. Ми робимо це тому, що функція dist () обчислює відстані за допомогою рядків.
довжина хвилі = dist (t (cluster_data))
довжина хвилі_clust = hclust (довжина хвилі, метод = «ward.d»)
ділянка (довжина хвилі _clust, зависання = -1, основний = «довжини хвиль, сильно пов'язані з міддю»)
прямого.hclust (довжина хвилі_clust, k = 2, яка = 2, межа = «синій»)
На малюнку нижче висвітлено скупчення довжин хвиль, найбільш сильно пов'язаних з поглинанням Cu 2 +.