5.15: Метагеноміка - вивчення мікробного світу
- Page ID
- 5786
Усі геноми, перелічені на моїй сторінці Розміри геному, описують повний геном одного виду. Для бактерій і археонів це означає, що організм був вирощений в чистій культурі, щоб забезпечити ДНК для секвенування. Але тепер зрозуміло, що мікробний світ містить величезну кількість обох груп, які ніколи не вирощувалися в лабораторії і, таким чином, уникли дослідження. Грунт, вода та вміст нашого товстого кишечника - приклади середовищ існування, які рясніють невідомими мікроорганізмами.
Завдяки нещодавній розробці секвенувальних машин, здатних швидко (і недорого) секвенувати величезну кількість ДНК, тепер практично послідовність ДНК, витягнутої зі складних мікробних екосистем, подібних до зразка ґрунту. Використовується кілька різних підходів, але всі залежать від першого етапу вилучення мікробної ДНК із зразка (та відділення її від набагато складнішої ДНК будь-яких еукаріотів, які можуть бути присутніми).
Оцінка мікробного різноманіття
ДНК, що кодує малу субодиницю (16S) рибосом як бактерій, так і археонів, містить деякі високо збережені області; тобто області ідентичної або майже однакової послідовності. Використовуючи праймери, які орієнтовані на ці регіони, можна отримати достатню кількість матеріалу за допомогою ПЛР полімеразної ланцюгової реакції для послідовності всього гена 16S рРНК.
Порівнюючи різні послідовності з базою даних послідовностей відомих організмів, можна оцінити, скільки різних типів мікробів присутня. Через значну генетичну різноманітність, виявлену між «штамами» одного виду (наприклад, E. coli K-12 та E.coli O157: H7), тісно пов'язані (> 97% ідентичності) послідовності 16S рДНК призначаються одному "філотипу", оскільки ми не можемо бути впевнені, чи належать вони до окремих видів або двох штамів одного виду. У будь-якому випадку колекція послідовностей 16S рДНК може бути організована таким чином, щоб сформувати філогенетичне дерево, щоб показати закономірності спорідненості.
Каталогізація генів в мікробній екосистемі
Аналіз генів 16S рДНК у зразку говорить нам, хто там, але, звичайно, не є повним геном і нічого не говорить нам про інші гени, присутні у різних членів популяції. Цю інформацію можна отримати шляхом «дробовика» секвенування зразка ДНК навколишнього середовища.
Кроки:
- Розбийте ДНК короткими фрагментами.
- Вставте їх у вектор, наприклад, плазміду, здатну рости в E. coli K-12.
- Піддайте клітини кишкової палички цій випадковій суміші і виростіть окремі бактеріальні клітини в колонії.
- Результат: бібліотека, що містить мільйони випадкових фрагментів ДНК з вихідного зразка.
- Виділяють плазміди і послідовно їх. Послідовність «читає» в середньому близько 100 нуклеотидів - набагато коротше, ніж ген, але часто досить часто, щоб перейти до наступного кроку.
- Використовуйте потужний комп'ютер, щоб спробувати зібрати фрагменти в лінійну послідовність ДНК. Комп'ютер шукає однакові розтяжки нуклеотидів у різних фрагментах і використовує перекриття, щоб зібрати їх у «контіг».
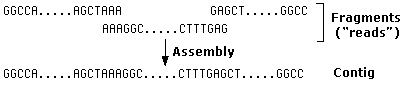
- Шукайте (шукайте комп'ютер) відкриті кадри читання (ORF) генів, кодуючих білок.
- Порівняйте ORF з даними відомих мікробів, які вже є в базах даних, щоб побачити, чи можна вивести функцію.
Сама різноманітність організмів у більшості мікробних екосистем робить практично неможливим знайти достатню кількість контигів, щоб зібрати повний геном для будь-якого організму, як ті, що перераховані в розмірах геному. Натомість ви отримуєте вікно у багато видів генів, присутніх у того чи іншого мешканця цієї екосистеми. Наприклад, ви можете виявити гени, які кодують білки, здатні деградувати забруднювачі навколишнього середовища або гени, здатні синтезувати новий антибіотик.
Пошук нових функцій у мікробних популяціях
Іншим способом використання метагеноміки є пошук нових функцій у господаря (наприклад, кишкової палички), якщо він може експресувати новий ген, з яким він був перетворений. Наприклад, скринінг бібліотеки клонів кишкової палички на здатність протистояти антибіотику може виявити гени, що беруть участь у резистентності до антибіотиків - тривожний розвиток останніх років.
Деякі області застосування метагеноміки
- Саргасове море: Метагеномний аналіз ДНК, витягнутої з морської води в Саргасовому морі, виявив наявність понад тисячі різних генів РДНК 16S (і, таким чином, приблизно такої кількості різних видів) та понад мільйон генів, кодуючих білок.
- Товста кишка людини: 0,3 г зразків фекалу від двох здорових людей вироблено 78 мільйонів базових пар послідовності. Кожен суб'єкт виробляв близько 25 тисяч відкритих кадрів для читання (ОРФ), з яких близько половини можна було б визнати вже відомими бактеріальними або архейними генами. Включені гени, що кодують ферменти для синтезу вітамінів (наприклад, вітаміну В 1), амінокислот та ферментів для перетравлення складних полісахаридів у нашому раціоні, які в іншому випадку були б неперетравлені. Можливо, цілих 10% енергії, яку ми витягуємо з нашої їжі, стає доступним для нас завдяки активності цих мікроорганізмів.
- Дренаж кислотних шахт: Метагеномний аналіз кислої води (рН ~ 0,5), що протікає з покинутої металевої шахти в Каліфорнії, виявив набагато простішу екосистему, ніж описані вище: лише 3 види бактерій та 2 архей. При такому обмеженому різноманітті вдалося зібрати майже повні геноми для двох цих організмів.
- Південноафриканський золотий рудник: Простіше все ще була екосистема, знайдена у воді 2,8 км (1,7 милі) вниз у золотому руднику. Звернувся тільки один організм: автотрофна бактерія, здатна витягувати енергію з неорганічних речовин в своєму середовищі і синтезувати з них всі необхідні для його життєдіяльності молекули.
